11月23日,《自然·方法》(Nature Methods)刊发刊发光电国家实验室生物医学光子学研究中心、生命科学与技术学院生物医学工程系曾绍群教授和龚辉教授研究组研究成果。该论文题为 《NeuroGPS-Tree:自动重建大规 模的密集神经元群落》(NeuroGPS-Tree: Automatic reconstruction of large-scale neuronal population with dense neurites)。
大脑是自然界最复杂的系统之一,支配着人类的一切活动。目前,人类对自身大脑的智能还所知不多,对老年痴呆、精神分裂等多种神经精神性疾病仍然 束手无策。解析人脑智能与疾病等需对从分子到整体有全面的认知。虽然科学家对宏观脑活动与微观的分子机制的认识,已有长足的进步,但是对介观层面的脑功 能,如细胞分辨的脑网络连接与功能等依然所知甚少。
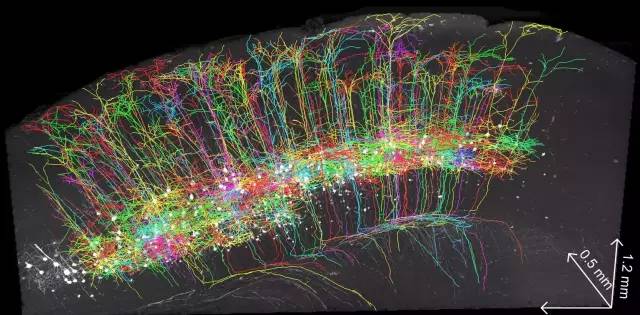
最近生命与信息科学与技术的融合发展,使得以细胞分辨脑网络的研究手段焕然一新,为破解这一难题 带来曙光。光学分子标记和显微成像技术的巨大进步,使得高分辨率获取小鼠全脑数据成为现实。然而如何从这些TB级海量数据中重建出真实的神经回路,已成为 严重制约能否将获取的数据转化为知识的瓶颈。特别是在类似于从茂密的森林图像中,如何准确地细分出一棵树上所有结构的形状,即如何从密集的神经群中重建单 神经元(数字化重建单神经元形态)在图像处理方面仍然是巨大的挑战。
光电国家实验室生物医学光子学功能实验室曾绍群教授、龚辉教授的研究组,借鉴人脑执行知觉功能时的先整体后局部的识别策略,递进式地利用多尺度 信息,快速地从神经元的三维荧光图像中重建出神经元群落,从而为绘制精细的脑功能图谱、重建大脑中的“ 信息高速公路”走出重要的一步。与以前方法主要专注 于重建单个神经元不同,这一工作专注于重建神经元群 落。通过对大量的神经元进行高效的定位、追踪、分配,在3小时内重建了960个神经元。
NeuroGPS- Tree方法首先用约束主曲线方法,追踪出所有的神经纤维,然后根据已有的神经元胞体位置和形状信息,用碰撞检测方法构建神经纤维之间、神经纤维与神经元 胞体之间的连接关系,最后将神经纤维分配到唯一对应的神经元中,从神经网络中分割出单个完整神经元。由于神经纤维分布的密集性,很多单神经元之间会出现虚 假的连接,导致神经纤维连成复杂交织的神经元网络而无法分割出单个神经元。该方法能识别并打断虚假的连接,从神经元网络中将每一个单神经元分解出来。受人 工分割神经元时人脑识别虚假神经纤维连接的启发,该方法将人脑识别虚假神经纤维连接的策略和神经元相关统计信息联合应用,利用神经网络多个尺度的神经纤维 连接信息,来识别并切断虚假连接,具有较高的准确性。
据介绍,“ 单细胞分辨全脑网络可视化”是生物医学光子学研究团队的重点研究方向。针对这一目标,该团队在2010年建立显微光学切片成像 (MOST)(Li et al. Science,2010)技术的基础上,发明了系列成像技术:神经元长程投射成像技术(Gong et al., Neuroimage, 2013),飞秒激光无惯性扫描双光子显微成像技术(Zheng et al., Optics Express, 2013),化学重激活荧光蛋白分子成像(Xiong et al., Nature Communications, 2014),并开发相应单细胞分辨脑网络大数据分析软件。此次的大规模神经网络重建是该团队在TB级图像大数据处理与重建方面的重大进展。相应软件可从 http://vbn.org.cn上下载。
此项工作由生物医学光子学研究中心、生命科学与技术学院生物医学工程系全廷伟、周航、李靖、李诗玮、李安安、李宇昕、吕晓华、骆清铭、龚辉(共 同通讯作者)、曾绍群(共同通讯作者)共同完成。曾绍群主要从事生物医学光学、生物医学成像、神经回路与蛋白质功能高分辨成像新技术新方法、生物信息技术 等研究。龚辉的主要研究方向包括认知光学成像与神经信息学、数字生命与医学影像。
该研究得到了科技部国家重大科学研究计划、国家自然科学基金、国家重大科学仪器设备开发专项、以及武汉光电国家实验室(筹)主任基金等项目的支持。
全文链接:http://www.nature.com/nmeth/journal/vaop/ncurrent/full/nmeth.3662.html
或扫描/长按下方的二维码获取全文:

(责任编辑:冀娴贤)