近日,华中科技大学武汉光电国家研究中心张智红教授团队在《Science China Life Sciences》上发表题为“C1M2: a universal algorithm for 3D instance segmentation, annotation, and quantification of irregular cells”的研究论文。目前,基于深度学习和特征工程的细胞分割方法在2D图像中取得了良好的表现,但对于3D图像中不规则细胞的准确分割仍是难题。本研究开发了一种针对不规则细胞的三维实例分割、标注和量化算法,称为Crop Once Merge Twice(C1M2),即切割一次、融合两次。研究结果表明,C1M2算法可以在没有细胞核图像的情况下,对厚组织3D图像中不规则细胞进行准确分割,进而获得准确的细胞三维形态、空间分布及分子表达信息,从而展示了其在组织流式中的应用潜力。
免疫荧光染色和成像是检测组织环境中细胞类型和蛋白质表达的常用技术,通常为5-10μm的薄组织切片。近年来发展的基于厚组织切片的高分辨三维荧光成像技术,可在组织结构信息更丰富的情况下获取细胞形态、空间分布、细胞间相互作用和分子表达信息,从而更好地理解生理病理环境下的细胞功能。然而,一些形态不规则的细胞在组织局部密集堆积(如血管周围的巨噬细胞和免疫器官内的树突细胞),细胞间的紧密接触使得细胞边界难以确定,严重降低了细胞分割算法的准确性,进而影响单个细胞计数和分子表达的定量分析。因此,准确、高效地分割厚组织三维图像中密集堆积的细胞,是研究复杂生理病理环境下组织结构和功能的前提。
在2D空间的细胞实例分割中,细胞的接触都存在于2D平面,可以很直观的在二维平面进行观察。而在三维空间内,不规则细胞之间的接触面具有不确定的空间方向,而这种空间方向很难寻找。针对此难点,研究人员开发了一种细胞实例分割算法:切割一次、融合两次(C1M2)。C1M2主要包含四个步骤。第一步,由于不规则细胞的细胞核通常很难定位,因此首先过分割细胞核,确保获得所有的细胞核。第二步,将粘连细胞切割为大小合适的块,块与块之间的缝隙包含有细胞之间和细胞内部的缝隙。第三步,进行第一轮融合,即以细胞核为基础,将块融合为实例细胞。细胞核是过分割,因此实例细胞也是过分割。不规则细胞通过细胞的触角相连,因此细胞与细胞之间的接触面积通常会小于细胞内部的接触面积。第四步,进行第二轮融合,即计算实例细胞的接触面积和体积,同时根据细胞的接触面积和体积将实例细胞从过分割状态融合为接近真实的状态,即具有小体积和大接触面积的两个细胞融合为一个细胞,反之则不融合两个细胞。经过两轮融合后,细胞内部的切割缝隙会被融合,而细胞之间的切割缝隙则会被保留,从而达到分割相连细胞的目的。两轮融合算法降低了对细胞核的依赖,因此研究人员可以从细胞的语义分割结果中计算得到伪细胞核,而不需要荧光染料特异性标记细胞核。
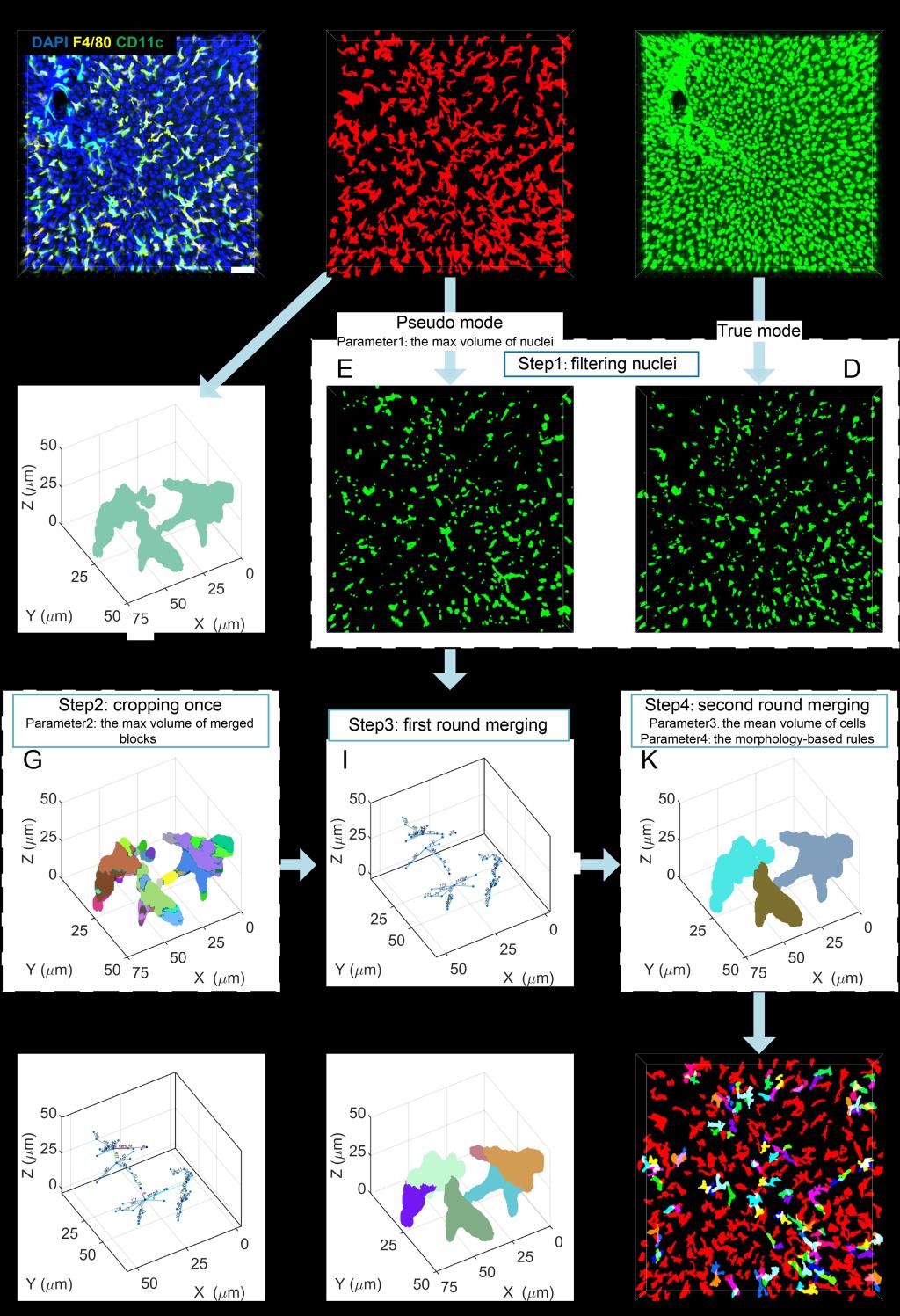
图1.实例分割算法流程图。(A)三维展示成像结果,图中绿色代表CD11c+细胞,黄色代表F4/80+细胞,蓝色代表所有细胞的细胞核。(B)F4/80+细胞的语义分割结果。(C)细胞核语义分割结果。(D)使用真核模式筛选后的细胞核。(E)使用伪核模式筛选的细胞核。(F)包含三个细胞的一个连通域。(G)将连通域切割为大小合适的块,每一个颜色代表切割的一个块。(H)将块结构转变为图结构。(I)第一轮融合将一个图结构分为5个子图。(J)根据图的节点编号将块进行融合。(K)第二轮融合结果。(L)对一幅3D图像的实例分割结果,红色代表独立细胞,其他颜色代表粘连细胞。
本研究采集了5套数据集进行算法测试。制备的样本和成像方法包含三种组织器官(肝脏、肾脏和脾脏),三种不规则细胞(CD11c+细胞、CX3CR1+细胞和F4/80+细胞)分别用内源性荧光蛋白和外源性荧光抗体标记,两种样本制作方法(厚组织切片和光透明方法),以及三种光学显微镜(蔡司710显微镜、Limo显微镜和光片照明显微镜)获取的三维图像。通过对现有方法的测试和对比,C1M2算法主要有三个优点:1)C1M2可以在过分割和欠分割中达到平衡,从而获得良好的准确率;2)通过开发的伪核模式,C1M2算法不需要细胞核就可以进行实例分割;3)C1M2具有很强的鲁棒性,改变成像的器官、细胞、样本制备方法和成像显微镜,C1M2均能获得良好的准确率。
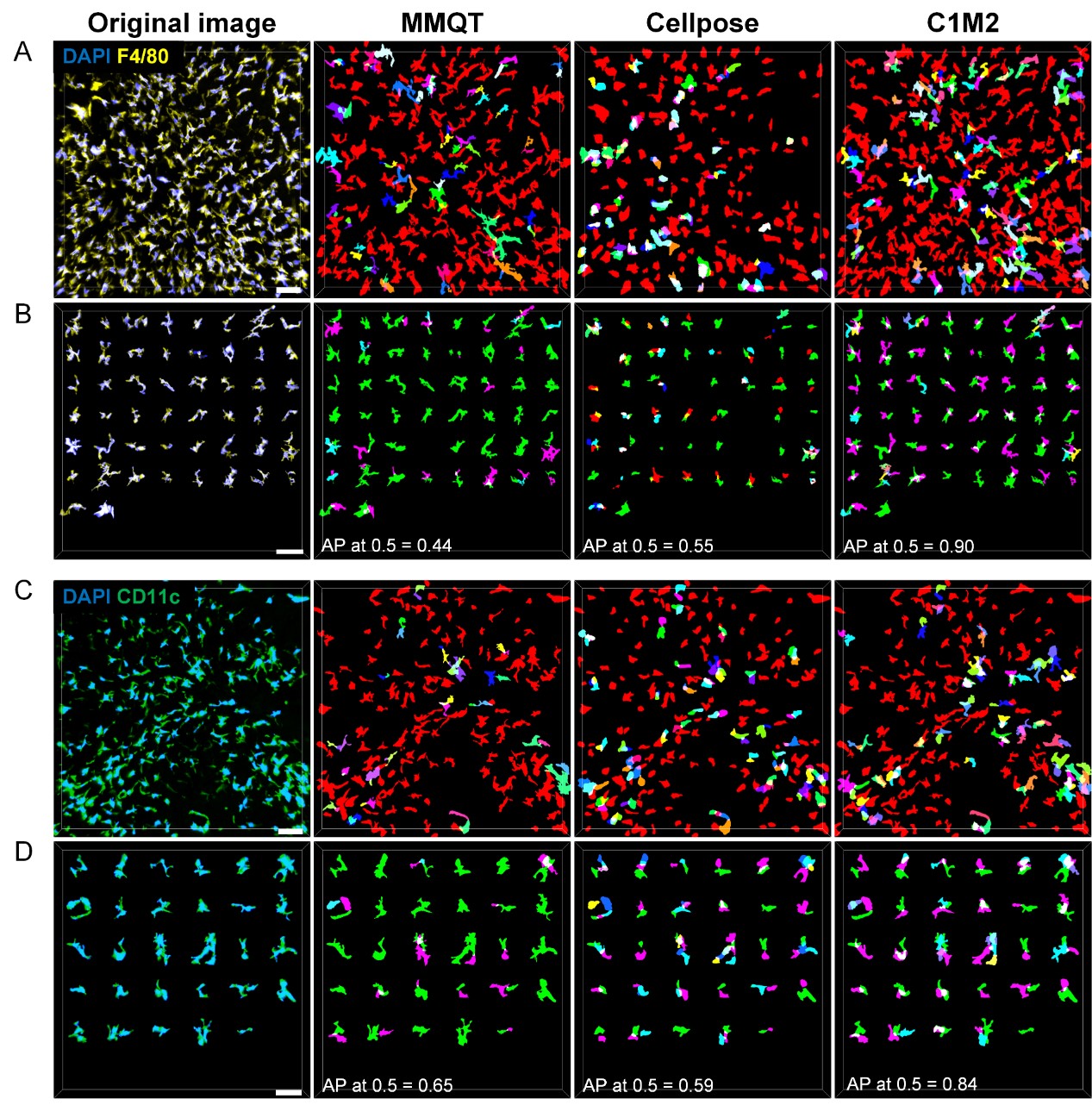
图2.对比三种算法在不同数据集上的结果。(A)在完整视野中对比MMQT、Cellpose和C1M2算法对于F4/80+细胞的分割结果,在原图中,黄色代表F4/80+细胞,蓝色代表细胞核。(B)图A中的粘连细胞。(C)在完整视野中对比MMQT、Cellpose和C1M2算法对于CD11c+细胞的分割结果,在原图中,绿色代表CD11c+细胞,蓝色代表细胞核。(D)图C中的粘连细胞。该数据对应表1中数据集1和2。

图3.使用C1M2算法的伪核模式分割不同器官中的CX3CR1+细胞。(A)红色为肝脏中CX3CR1+细胞的语义分割结果,黄色为使用伪核模式检测到的细胞核,该数据对应表1中数据集3。标尺:100μm。(B)C1M2算法的实例分割结果,红色为孤立细胞,其他C1M2分割的粘连细胞。(C)红色为脾脏中CX3CR1+细胞语义分割结果,黄色为使用伪核模式检测到的细胞核。标尺:200 μm,该数据对应表1中数据集4。(D)脾脏中CX3CR1+细胞的实例分割结果,红色为孤立细胞,其他颜色为粘连细胞。(C)红色为肾脏中CX3CR1+细胞语义分割结果,黄色为使用伪核模式检测到的细胞核,该数据对应表1中数据集5。标尺:300 μm。(D)肾脏中CX3CR1+细胞的实例分割结果,红色为孤立细胞,其他颜色为粘连细胞。
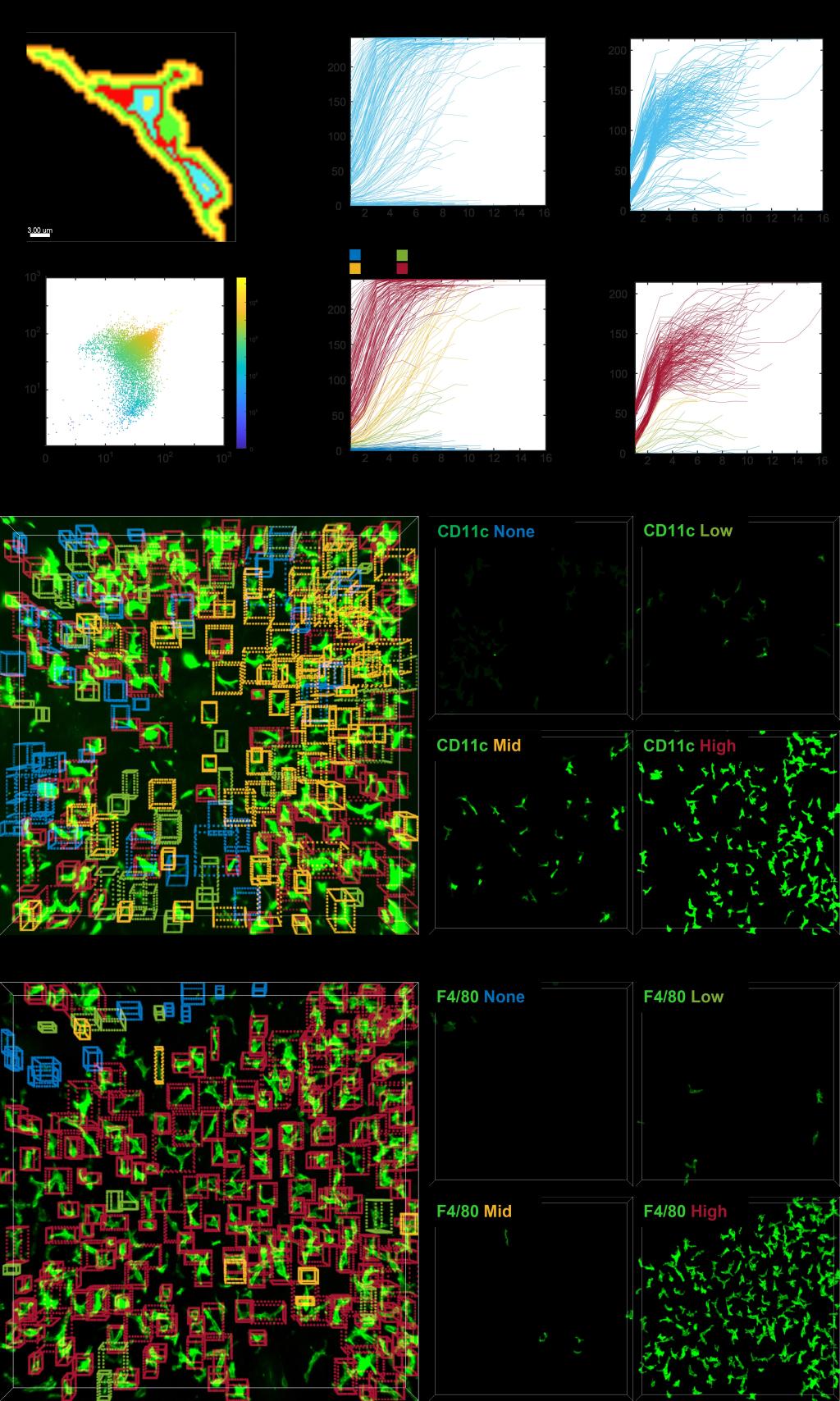
图4.可视化方法原位展示细胞荧光强度量化和标注结果。(A)2D展示不同半径的细胞环,其中不同颜色代表不同半径的细胞环。(B)细胞环对应的CD11c通道的荧光强度,每一条曲线代表一个细胞,其中纵轴为细胞环的荧光强度,横轴为细胞环半径。(C)细胞环对应的F4/80通道的荧光强度。(D)将两个细胞环对应的F4/80和CD11c荧光强度进行合并。(E)F4/80+细胞自动标注结果,每种颜色的矩形框代表一种表达水平,其中蓝色代表无表达、绿色代表低表达、黄色代表中表达和红色代表高表达。(F)CD11c+细胞自动标注结果。
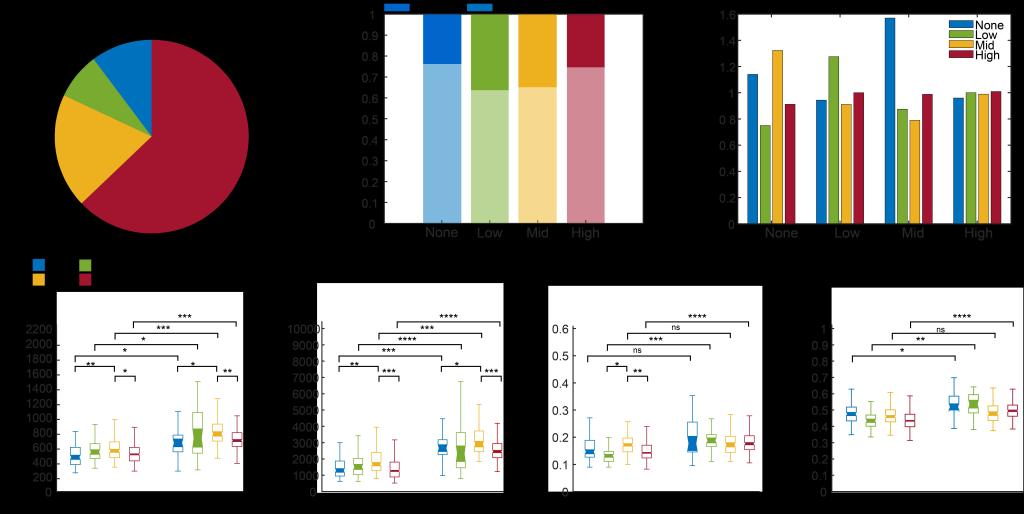
图5. F4/80+细胞量化分析结果。(A)F4/80+细胞的四种分子表达水平的细胞比例。(B)在每种分子表达水平的F4/80+细胞中,孤立细胞和粘连细胞的比例。(C)每个细胞与其距离最近细胞的类别比例。(D)F4/80+细胞的四种形态学量化参数。组间差异为ns表示没有显著性差异,*表示P < 0.05,**表示P < 0.01,***表示P < 0.001,****表示P < 0.0001。
细胞的形态和功能具有一定的相关性,在厚组织原位直观可视化地显示单个细胞的形态和特定分子的表达量具有重要意义。流式细胞术和单细胞RNA测序技术可以测量细胞内的分子表达水平,实现对单个细胞的定量分析与分类。但是这两种方法需要先将组织进行酶解消化成单细胞悬液,此过程会丢失细胞的空间信息和形态学信息。厚组织切片3D图像内的细胞荧光强度的三维量化分析难点在于:当细胞与细胞粘连时,两个粘连的细胞容易被判定为一个细胞,由此得到的细胞空间位置和形态学信息是不准确的。同时,如果两个粘连细胞的抗原表达丰度不一致时,所获取的分子表达量信息也是错误的。基于C1M2算法处理后的精确3D细胞实例分割结果,研究人员开发了一套自动量化和标注荧光强度的算法。其中,自动量化算法能够在3D空间上将细胞分为半径不同的同心细胞环,每个细胞环的形态和最外层细胞表面保持一致,并使用3D同心细胞环测量对应细胞位置上的荧光强度;自动标注算法通过计算荧光强度和细胞环半径的关系,将荧光抗体的表达量自动标注为4种水平,包括无表达、低表达、中表达和高表达。通过该算法,研究人员可以同时获得细胞形态学信息、空间位置信息和分子表达水平信息。进而,配合商业软件Imaris制作了可视化软件,在厚组织原位可视化展示细胞的分子表达水平、细胞空间位置和细胞形态。
综上所述,研究人员开发了一种针对不规则细胞的三维实例分割算法C1M2,并验证了该算法在多种数据集中的准确性。进而,基于精确的3D细胞实例分割结果,开发了一套自动量化和标注荧光强度的算法,并且能够在原位可视化展示细胞的分子表达水平、细胞空间位置和细胞形态等信息,从而实现组织流式的准确定量分析功能。
该研究得到国家重点研发计划(2017YFA0700403,2017YFA0700402)、国家自然科学基金(62061160490)、武汉应用基础研究(2020010601012167)、中央高校基本科研业务费专项基金(2019kfyXMBZ022)和武汉光电国家研究中心创新基金的资助。华中科技大学武汉光电国家研究中心张智红教授和电子信息与通信学院杨欣教授为本文的通讯作者,课题组博士研究生郑好和博士后黄松林为文章的共同第一作者,博士研究生张静、章仁、王佳璐,以及武汉光电国家研究中心李安安教授和袁菁教授共同参与了相关工作。感谢武汉光电国家研究中心全廷伟副教授对本研究的指导和帮助。
全文链接:http://engine.scichina.com/doi/10.1007/s11427-022-2327-y
代码链接:https://github.com/zhengbetter/C1M2